产品展示
优质供应
详细内容
文献:Targeted in situ genome-wide profiling with high efficiency for low cell numbers

随着技术的发展,ChIP-seq的改进可以实现TFs 6-8碱基对分辨率的映射,但实验结果还是存在很多问题,比如说ChIP的高背景限制了灵敏度,需要的细胞比较多以及交联和增溶产生的假象。而CUT&RUN相比于传统ChIP在细胞量和信噪比等方面的优势。
1)在整个实验过程中,pMNase的量对片段化的结果至关重要
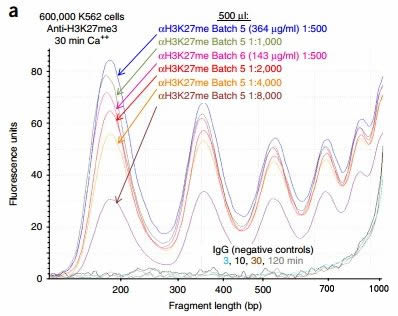
fig1.两批样品500μl体积中,600,000个K562细胞,加入不同浓度的pA-MN,片段化之后,采用TapeStation 通过分析荧光强度来分析pA-MN对H3K27me3基因组片段化的影响。
2)CUT&RUN技术用于少量细胞,信噪比很好
研究者检测了100-6000个K562细胞的H3K27me3的基因组分布,发现在低至100细胞时,CUT&RUN仍可以很好地检测出H3K27me3的峰,而且在异染色质区也样可以看到峰。
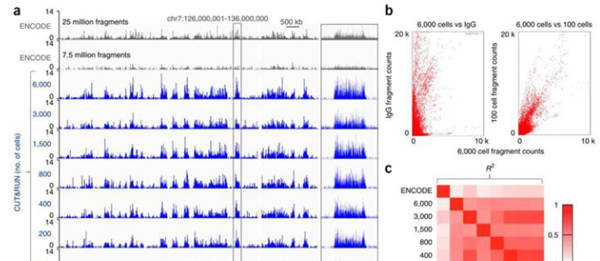
fig2.CUT&RUN可以检测低至100细胞的H3K27me3基因组分布(a)ENCODE数据库中的K562细胞的H3K27me3 ChIP-seq结果,及6000-100个K562细胞的H3K27me3 CUT&RUN结果。(b)CUT&RUN结果的散点图,以50bp为个bin,相对于阴性对照IgG,6000细胞与100细胞有明显更强的相关性。(c)热图显示6000-100细胞的CUT&RUN结果有很好的相关性。
3)CUR&RUN技术检测的信噪比要比传统ChIP-seq高很多
研究者检测了转录因子CTCF的基因组结合位点,发现CUR&RUN技术检测比ENCODE的CTCF ChIP-seq的信噪比要高很多,而且只需1.25万细胞就能得到很高质量的结果。尽管用1000细胞则会损失些峰。
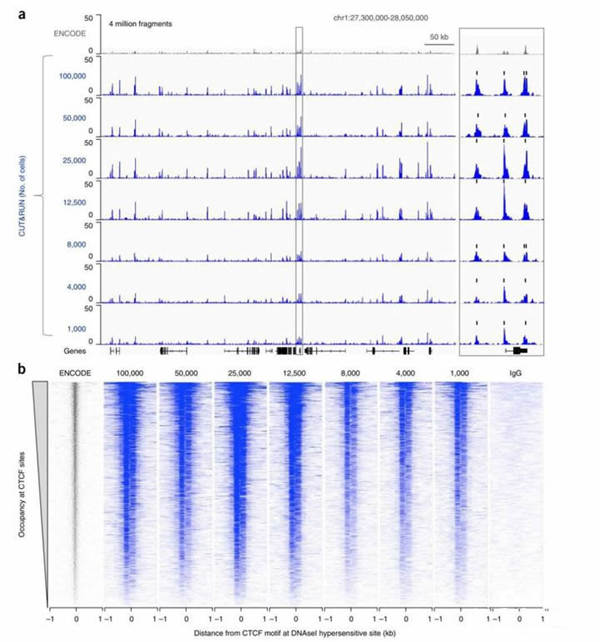
fig3.用CUT&RUN检测CTCF的结合位点,比ENCODE中的CTCF ChIP-seq信噪比高很多。
本文图2、图3分析源自CST公司。
艾美捷作为表观遗传域业的解决方案供应商,推荐EpiGentek的EpiNext CUT&RUN Fast Kit,cat#:P-2028,该试剂盒从低输入提供无超声破碎,以可靠地识别真正的目标蛋白质富集区域并实现高分辨率定位。可满足您快速富集蛋白质结合的DNA并绘制全基因组蛋白质/DNA相互作用的图谱的需求。










